Profesor Tiempo Completo – Departamento de Bioingeniería
El descubrimiento de las enzimas de edición genética es un avance tecnológico que tiene un impacto en diversas áreas del conocimiento, siendo la gran promesa para desarrollar métodos eficientes de terapia genética [1]. En este sistema el RNA guía (gRNA) permite un reconocimiento específico del sitio de corte en el DNA por las nucleasas de la familia CRISPR como la enzima Cas9. Sin embargo, se desconocen los mecanismos por los cuales, en ciertas circunstancias, Cas9 corta el DNA a pesar de reconocer regiones inespecíficas no complementarias o no-homologas. Se han desarrollado versiones mutantes de Cas9 que poseen buena discriminación de fragmentos no-homólogos, y evitan cortes fuera de contexto. No obstante, estas versiones muestran una eficiencia de corte disminuida [2]. A pesar de los numerosos reportes del sistema CRISPR, aun no se ha dilucidado los fundamentos de la activación de la nucleasa Cas9 cuando produce cortes inespecíficos en el DNA fuera de contexto (“off-target”).
La crio-microscopia electrónica (CME) permite obtener vistas de la interacción entre la enzima Cas9, el RNA guía (gRNA) y la interacción del dúplex gRNA-DNA con Cas9. CME muestra estructuras de interacción a nivel molecular. Por primera vez, la técnica de CME es empleada para dilucidar la cinética de interacción molecular de la nucleasa Cas9 en su activación para realizar cortes durante en un evento inusual de apareamiento no-homologo [3].
Se sabe que los dominios HNH y RuvC de Cas9 son los responsables del corte del DNA. Imágenes por CME muestran que la interacción molecular de una región RuvC es la responsable de escanear la interacción gRNA-DNA para detectar apareamientos no-homólogos. Mas aun, RuvC suele estabilizar eventos de dúplex gRNA-DNA en presencia de apareamiento no-homologo. RuvC mediante su dominio “loop” erróneamente activa a la enzima reorganizando sus regiones de corte a pesar de que existen nucleótidos no apareados en el dúplex gRNA-DNA [3]. ¿Qué ocurre si los residuos que en RuvC encargados de escanear al dúplex y activar a la nucleasa Cas9 son mutagenizados? Si la mutagénesis dirigida por CME logra obtener versiones mutantes de Cas9 que previenen un reconocimiento inespecífico sin afectar su eficiencia de corte seria un gran avance para el empleo de la edición genética de una manera más segura.
Las aplicaciones terapéuticas de precisión son la gran promesa para el aprovechamiento del sistema CRISPR-Cas9. Para que esta metodología pueda ser empleada rutinariamente, es necesario minimizar los riesgos de cortes inespecíficos en el genoma. Se han desarrollado variantes de Cas9 llamadas de “alta fidelidad” que minimizan los cortes fuera de contexto. En estas versiones iniciales de Cas9, la eficiencia de corte o edición genética es mucho más baja [4].
El primer paso es determinar las posiciones de los nucleótidos no-apareados en el dúplex gRNA-DNA. Las imágenes CEM identificaron que cuando los nucleótidos no-apareados se encuentran a una distancia de 18-20 posiciones respecto de la secuencia PAM, causan una tasa de reducción en el corte del DNA de 40 veces. Nucleótidos inespecíficos localizados en posiciones cercanas a PAM tienen una menor posibilidad de activar a Cas9 en un evento de apareamiento no-homologo RNA-DNA. En cualquiera de estos casos solo el 80% de estos
sustratos se logra cortar en un lapso de 1 hora, al incubar Cas9 con gRNA-DNA in vitro. Sabiendo que en condiciones normales la eficiencia de corte in vitro de Cas9 es de 1 segundo. Por lo tanto, los nucleótidos no-homólogos en las posiciones distales como 18-20 posiciones respecto de la secuencia PAM, suelen escapar la vigilancia y activar a Cas9 causando un corte fuera de contexto. Los nucleótidos no-apareados se encuentran mas cerca de PAM tienen más probabilidades de no activar a Cas9.
Las estructuras crio-microscópicas mostraron que el extremo distal del dúplex gRNA-DNA poseen una conformación lineal relativa próximo a PAM, ese estado difiere del determinado previamente por la versión Cas9 con interacción homologa 100% que poseen un ángulo típico de 70° (Figura 1). Esto significa que una posición típica se adopta con apareamientos no-homólogos distales (Figura 1, 18-20, 1 min) y difícilmente es detectado apareamiento fallido y logra activar a Cas9. En cambio, una posición de apareamiento no-homólogos cercano a PAM es atípica y es detectada por su conformación (Figura 1, 12-14, 5 min y 60 min) y no se activa Cas9.
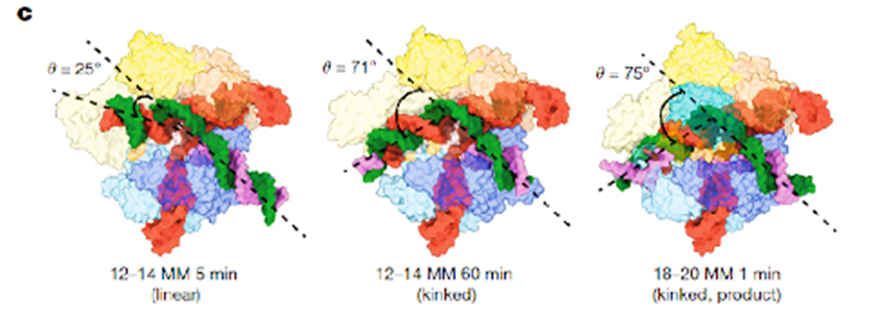
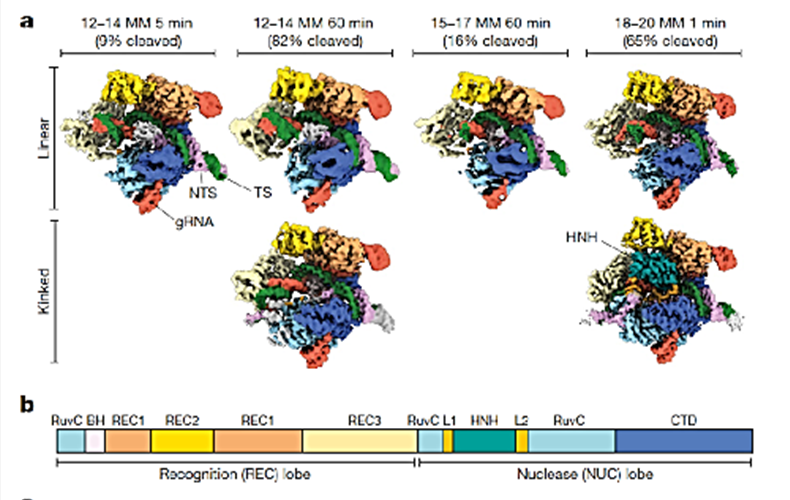
En la Figura 1, se aprecian dos conformaciones diferentes: un dúplex lineal y el dúplex con un ángulo de 70°. Las conformaciones de Cas9 en ambos casos son idénticas, pero el dúplex gRNA-DNA distal a PAM tiene un recorrido de 30 Å y se ancla con el dominio REC3 de la nucleasa. Se plantea la hipótesis de que los nucleótidos no-homólogos que previenen la interacción gRNA-DNA distal con REC3 pueden inducir una conformación no canónica lineal en lugar de producir el doblamiento de 70° normal en el dúplex, reduciendo la eficacia de corte. Por lo tanto, se postula como modelo que se produce un dúplex lineal gRNA-DNA que precede a la conformación de 70° requerido para activar el corte por Cas9. Los nucleótidos no-homólogos en posiciones distantes a PAM (18-20), pueden bloquear la conformación de 70° y escapan de la vigilancia por el dominio REC3, se activa Cas9 y el DNA es cortado (Figura 2).
¿Como algunas secuencias de DNA que no tienen homología perfecta son cortadas por Cas9? ¿Cómo evaden unos nucleótidos el mecanismo de control molecular de Cas9 y no otros?
Luego de la incubación de Cas9 con el DNA con nucleótidos no-homólogos en la posición 18-20, al minuto de la incubación, el 65% del DNA ha sido cortado. Las observaciones crio-microscópicas de estas estructuras permitieron identificar moléculas lineales y especies con un doblamiento normal de 70° requerido para activar el corte por Cas9. En las especies con doblamiento, el dominio HNH se posiciona en su conformación activa. Esto indica que las moléculas lineales forman parte de un primer evento que posteriormente cambia a un doblamiento que induce el anclaje de HNH y gRNA-DNA. Estos dos estadios, el lineal y de doblamiento permitieron descubrir el rol de las regiones “linkers” L1 y L2 que facilitan la interacción de los dominios de corte HNH y RuvC, respectivamente. Las versiones de enzimas Cas9 de alta precisión como “hyper-accurate-Cas9” y la versión Cas9 de alta fidelidad (Cas9-HF1), ambas con 4 mutaciones puntales, minimizan los cortes inespecíficos, pero se reduce la eficiencia de edición genética. Los residuos mutagenizados se localizan en el dominio REC3 que realiza interacción activa con el dúplex de 70°. Las mutantes, por lo tanto, tienen una menor capacidad de estabilizar el dúplex y L1 no puede tan fácilmente posicionar a HNH para el corte por lo que la eficiencia de edición es afectada. En estas versiones de alta fidelidad se reduce la activación de Cas9 en DNA no-homologo inespecíficos, pero también afecta el corte en los sitios específicos.
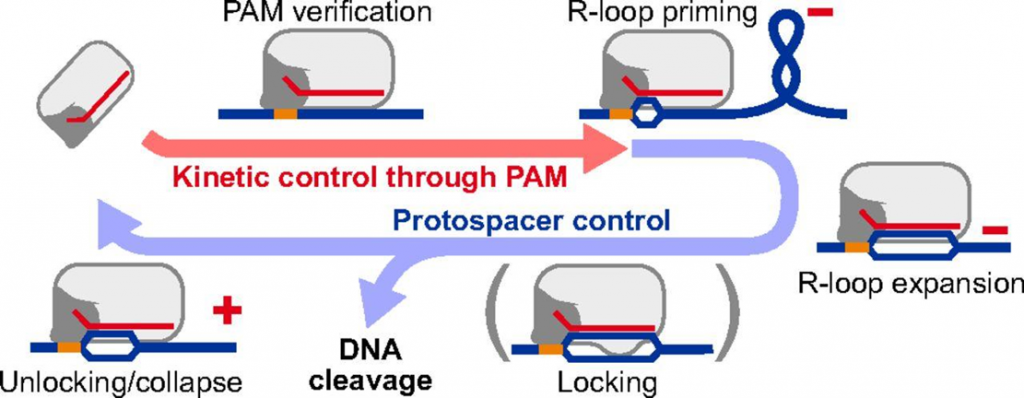
El modelo de R-loop para la función de nucleasas Cas propone que la enzima aproxima al gRNA creando una burbuja inicial en el DNA la cual se expande para lograr el apareamiento del gRNA y el anclaje de las regiones HNH y RuvC (Figura 3). La crio-ME permitió identificar a los residuos de aminoácidos que interactúan con la región R-loop, que son los que promueven la estabilidad de la proteína aun en presencia de nucleótidos no-homólogos a diferencia de las versiones de alta fidelidad, dejando intactas las regiones REC3, L1 y L2 permitiendo posicionar a los dominios de corte HNH y RuvC. Por lo tanto, la nueva versión SuperFi-Cas9 posee 7 mutaciones en el “loop” de RuvC responsable de la tolerancia a secuencias no-homologas. Se cree que esta región en RuvC estabiliza el dúplex aun en presencia de nucleótidos no-homólogos activando a Cas9 para realizar cortes fuera de contexto.
Las mutaciones RuvC en esta región no afectan la eficiencia de corte en el caso de homología perfecta, pero no estabilizan al dúplex en caso de apareamiento no-homologo. Por lo tanto, esta versión de la enzima llamada SuperFi-Cas9, desarrollada exclusivamente con la poderosa herramienta de crio-microscopia electrónica permite una edición genética en sitios homólogos y evita cortes inespecíficos fuera de contexto. La crio-microscopia muestra el potencial para mejorar los sistemas de edición genética que deben ser empleados en técnicas de diagnóstico y reparación de genes defectuosos y cuyo empleo se ha visto limitado por los cortes fuera de contexto que podría causar efectos nocivos en las células. La técnica CME va a permitir obtener una nueva generación de “tijeras moleculares” mejoraras y más confiables para sus aplicaciones en Biomedicina [3,4].
Referencias
1.- Doudna JA. The promise and challenge of therapeutic genome editing. Nature. 2020 Feb;578(7794):229-236. doi: 10.1038/s41586-020-1978-5. Epub 2020 Feb 12. PMID: 32051598; PMCID: PMC8992613. Doudna JA.
2.- Kleinstiver BP, Pattanayak V, Prew MS, Tsai SQ, Nguyen NT, Zheng Z, Joung JK. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 2016 Jan 28;529(7587):490-5. doi: 10.1038/nature16526. Epub 2016 Jan 6. PMID: 26735016; PMCID: PMC4851738.
3.- Bravo, J.P.K., Liu, MS., Hibshman, G.N. et al. Structural basis for mismatch surveillance by CRISPR–Cas9. Nature 603, 343–347 (2022). https://doi.org/10.1038/s41586-022-04470-1
4.- Kim N, Kim HK, Lee S, Seo JH, Choi JW, Park J, Min S, Yoon S, Cho SR, Kim HH. Prediction of the sequence-specific cleavage activity of Cas9 variants. Nat Biotechnol. 2020 Nov;38(11):1328-1336. doi: 10.1038/s41587-020-0537-9. Epub 2020 Jun 8. PMID: 32514125.