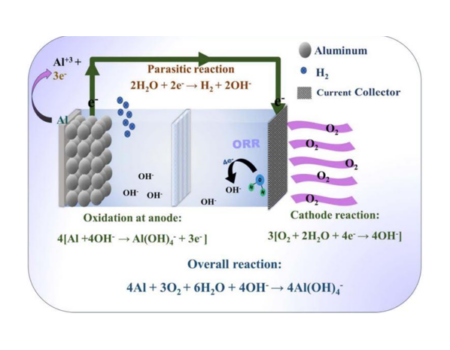
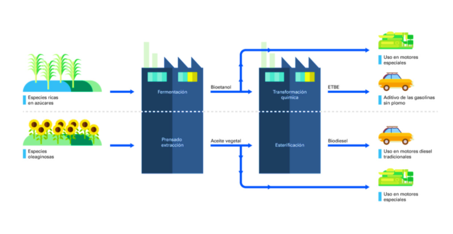
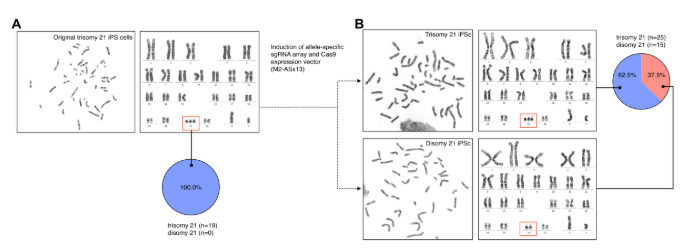
Profesor a Tiempo Completo
Dpto. de Bioingeniería e Ingeniería Química

El bloqueo de puntos de control inmunológico dirigido a PD-1 (Programmed Cell Death Protein 1) muestra un gran éxito en la terapia contra el cáncer. Sin embargo, el mecanismo de cómo la unión del ligando (PD-L1 o PD-L2) inicia la señalización de PD-1 aún no está claro. Como marcador de pronóstico de múltiples cánceres, el PD-L1 soluble se encuentra en el suero de los pacientes y puede unirse al PD-1, pero no logra suprimir la función de las células T. Previamente se demostró que las células T ejercen fuerzas endógenas sobre los enlaces PD-1-PD-L2 e impulsan la hipótesis de que la fuerza mecánica podría ser crítica para la activación de PD-1. En el caso del ligando soluble, no existe un fuerza mecánica debido a la falta del soporte mecánico proporcionado por ligando anclado a la superficie.
Un estudio reciente (Li K, Cardenas-Lizana, and et al.) ha revelado que la interacción entre PD-1 y sus ligandos está significativamente influenciada por la fuerza mecánica generada al momento de la unión que es un aspecto fascinante del PD-1. El estudio muestra que la función PD-1 se elimina o reduce cuando se elimina o humedece el soporte mecánico del ligando, respectivamente. PD-1 es una proteína que se encuentra en las células T, que son componentes esenciales del sistema inmunológico. Actúa como un punto de control inmunológico que ayuda a regular las respuestas inmunes y prevenir la autoinmunidad. Cuando PD-1 se une a su ligando, PD-L1, puede inhibir la actividad de las células T, promoviendo la muerte celular programada en ciertas células T, particularmente aquellas que se activan contra antígenos específicos.
Los Catch bonds and slip bonds son respuestas moleculares que describen dos comportamientos diferentes de la unión entre receptores y ligandos cuando se someten a una fuerza mecánica. Slip bond es un enlace cuya vida útil disminuye al aumentar la fuerza aplicada. Este es el comportamiento más intuitivo, ya que una mayor fuerza normalmente desestabiliza los enlaces. El Catch bond es un enlace cuya vida útil aumenta con la fuerza aplicada. Este comportamiento contrario a la intuición es esencial en muchos procesos biológicos, como la adhesión celular.
Resultados claves:
- Catch Bonds: bajo fuerzas bajas (menos de 7 pN), PD-1 forma Catch Bonds con sus ligandos, lo que significa que la fuerza del enlace aumenta con la fuerza aplicada. Esta vida útil prolongada del vínculo podría potencialmente mejorar las señales inhibidoras inmunitarias.
- Slip Bonds: las fuerzas superiores a 8 pN transforman los enlaces del ligando PD-1 en Slip Bonds, donde la fuerza del enlace disminuye al aumentar la fuerza, lo que lleva a una disociación más rápida y generando cambios estructurales inducidos por fuerzas.
- Implicaciones funcionales: la capacidad de las células para «sondear» el entorno mecánico a través de interacciones PD-1 sugiere un mecanismo novedoso para regular las respuestas inmunes basadas en señales físicas.
La inmuno-Ingeniería es un campo interdisciplinario que combina principios de ingeniería con inmunología para diseñar soluciones innovadoras para el tratamiento de enfermedades. Al comprender las complejidades del sistema inmunológico, los investigadores pueden desarrollar terapias, diagnósticos y materiales novedosos para mejorar la función inmunológica o redirigirla hacia objetivos específicos. Esta investigación abre nuevas vías en la inmuno-Ingeniería para aplicaciones de la inmunoterapia. Manipular la función moduladora de PD-1 mediante el cambio del entorno mecánico podría suprimir o mejorar la señalización de PD-1. Asistir en el diseño de nuevas terapias mediante el desarrollo de moléculas que puedan interferir con los cambios inducidos por la fuerza en PD-1 y así conducir a mejores terapias contra el cáncer. Ayudar en la comprensión de las propiedades mecánicas del microambiente tumoral que podrían influir en la función de las células inmunitarias a través de interacciones PD-1. Investigar a profundidad los cambios estructurales inducidos por la fuerza en PD-1 y sus ligandos podría proporcionar una comprensión más completa de los mecanismos subyacentes.
Esto ayudaría en la exploración del potencial terapéutico de la función modular de PD-1 a través de medios mecánicos, como el uso de nanopartículas o ultrasonido. Finalmente, se podría investigar el rol de las fuerzas mecánicas en la regulación de la función de PD-1 directamente modelos animales de cáncer. Actualmente profesores de Bioingeniería de UTEC se encuentran trabajando en proyectos relacionados a esta investigacion. Si estás interesado en ser parte de los proyectos que esperas: ¡Únete a Bioingeniería de la UTEC!

References:
- Li K, Cardenas-Lizana P, Kellner AV, Yuan Z, Ahn E, Lyu J, Li Z, Salaita K, Ahmed R, Zhu C. Mechanical force regulates ligand binding and function of PD-1. bioRxiv [Preprint]. 2023 Aug 15:2023.08.13.553152. doi: 10.1101/2023.08.13.553152. PMID: 37645980; PMCID: PMC10462004.
- Zhu C., Chen W., Lou J., Rittase W. & Li K. Mechanosensing through immunoreceptors. Nature immunology 20, 1269–1278 (2019).
- Liu B., Chen W., Evavold B. D. & Zhu C. Accumulation of Dynamic Catch Bonds between TCR and Agonist Peptide-MHC Triggers T Cell Signaling. Cell 157, 357–368 (2014).
- Choi H.-K., Cong P., Ge C., Natarajan A., Liu B., Zhang Y., Li K., Rushdi M., Chen W., Lou J., Krogsgaard M. & Zhu C. Catch bond models may explain how force amplifies TCR signaling and antigen discrimination. Nature communications (2023).
- Li K., Cheng X., Tilevik A., Davis S. J. & Zhu C. In situ and in silico kinetic analyses of programmed cell death-1 (PD-1) receptor, programmed cell death ligands, and B7–1 protein interaction network. The Journal of biological chemistry 292, 6799–6809 (2017).
- Liu B., Chen W. & Zhu C. Molecular force spectroscopy on cells. Annu Rev Phys Chem 66, 427–451 (2015).
- Zhu C., Chen Y. & Ju L. A. Dynamic bonds and their roles in mechanosensing. Curr Opin Chem Biol 53, 88–97 (2019).
- Chesla S. E., Selvaraj P. & Zhu C. Measuring Two-Dimensional Receptor-Ligand Binding Kinetics by Micropipette. Biophys J 75, 1553–1572 (1998).
- Chen W., Zarnitsyna V. I., Sarangapani K. K., Huang J. & Zhu C. Measuring Receptor-Ligand Binding Kinetics on Cell Surfaces: From Adhesion Frequency to Thermal Fluctuation Methods. Cell Mol Bioeng 1, 276–288 (2008).